2. CHAPITRE 2 Prise en main et manipulation des données▲
Objectifs : Ce chapitre permet d'acquérir quelques bases dans la manipulation de données sous R. Il permet d'abord de se familiariser avec les principaux objets (vecteurs, matrices, tables) et les opérations les plus courantes (opérations, assignations, sélections, tests conditionnels, recodages).
Prérequis Notions de base du fonctionnement de R et de l'interface RStudio, telles que présentées dans le Chapitre 1CHAPITRE 1 Prise de contact.
Description des packages utilisés Les manipulations effectuées dans ce chapitre nécessite l'utilisation de deux packages :
- Package sqldf : il permet d'exécuter des requêtes SQL (Structured Query Language) sur les tableaux. Ce package est constitué d'une unique fonction éponyme, qui prend pour argument la requête SQL souhaitée.
- Package reshape2 : il propose plusieurs fonctions utiles pour manipuler et reformater les tableaux de données.
- Package plyr : il sert principalement à diviser un objet en blocs, appliquer une fonction à chaque bloc et recoller les résultats obtenus pour chaque bloc (split-apply-combine strategy). Il propose également quelques fonctions simples pour recoder et trier.
- Package dplyr : ré-écriture récente de plyr, ce package très prometteur simplifie la plupart des manipulations présentées dans ce chapitre et en améliore la vitesse d'exécution.
2-1. Description et manipulation des objets▲
R est un langage orienté objet : tout ce qui est créé et manipulé sous R est un objet. Ces objets permettent de stocker et de structurer les données.
2-1-1. Les principaux types d'objets▲
- Vecteur (vector) : suite unidimensionnelle et ordonnée de valeurs. Les trois principaux types de vecteurs sont : numeric (entier ou double précision), character (alphanumérique), boolean (TRUE/FALSE).
- Facteur (factor) : vecteur qui ne peut prendre qu'un nombre fini de modalités prédéclarées. Ce vecteur est défini par des niveaux (levels), qui sont les valeurs effectivement prises par la variable, et d'étiquettes (labels), qui sont associées aux niveaux. Par exemple, une variable définissant le sexe des individus pourrait être codée avec les entiers 1 et 2 (levels) auxquels correspondraient les étiquettes (labels) Homme et Femme. Plusieurs fonctions de création de tableaux ou d'importation de données transforment automatiquement les champs alphanumériques en facteurs.
- Matrice (array ou matrix) : un objet array est une matrice, c'est-à-dire un vecteur multidimensionnel. Un objet matrix est un sous-type de l'objet array, c'est une matrice bi-dimensionnelle. Les données stockées dans une matrice sont nécessairement d'un seul et même type : booléen, entier, alphanumérique, etc. Avec RStudio, dans la fenêtre Environment on peut cliquer sur les objets de type matrice et les afficher.
- Liste (list) : liste ordonnée d'objets. Une liste permet de regrouper un ensemble d'objets de tailles différentes et de natures diverses : vecteurs, matrices ou tout type d'objet existant dans R. Par exemple, on peut stocker l'ensemble des résultats d'un modèle de régression dans une seule et même liste qui contiendrait un vecteur numérique de longueur n de valeurs espérées, un vecteur numérique de longueur n de résidus, un vecteur numérique de longueur 1 stockant une p-value, un vecteur booléen sur la significativité du résultat, etc.
- Tableau (data.frame) : tableau de données. Quand on importe un fichier depuis un tableur ou depuis un format SAS ou SPSS, l'objet créé est par défaut un data.frame. Pour les utilisateurs de tableurs et de logiciels d'analyse comme SAS ou SPSS, le data.frame sera le type d'objet le plus familier. Cet objet tableau combine des caractéristiques de liste et de matrice : c'est une liste de vecteurs assortis d'un nom (intitulé de colonne). À la différence d'une matrice, les vecteurs contenus dans le tableau peuvent être de différentes natures : valeurs numériques, alphanumériques, booléennes. À la différence d'une liste, les vecteurs contenus dans le tableau doivent être de même taille. Avec RStudio, dans la fenêtre Environment on peut cliquer sur les tableaux et les afficher.
- Autres objets : il existe une grande diversité de types d'objets du fait que certaines fonctions créent des objets sur mesure. La fonction lm() (linear model) créé par exemple un objet de type lm qui est une liste d'objets (fitted.values, p.value, etc.). Les données spatiales sont aussi stockées dans des objets particuliers qui sont détaillés au chapitre correspondant (cf. Chapitre 10CHAPITRE 10 Introduction aux objets spatiaux et à la cartographie).
- Fonction : deux caractéristiques importantes des fonctions doivent être soulignées, d'une part les fonctions sont des objets au même titre que les vecteurs ou les matrices, d'autre part les fonctions 14 Prise en main et manipulation des données peuvent prendre d'autres fonctions comme argument. Ces aspects sont détaillés dans le Chapitre 3CHAPITRE 3 Introduction à la programmation.
L'existence de différents types d'objets peut être déroutante mais elle devient une richesse lorsqu'on sait s'en servir. Le pendant de cette diversité est qu'il faut régulièrement se soucier du format des objets et les transformer d'un type à un autre.
2-1-2. Le vecteur et le facteur▲
Avant de présenter les principales méthodes de création et de manipulation des objets, un bref focus s'impose sur deux types d'objets fondamentaux : le vecteur et le facteur. Un vecteur est une suite ordonnée de valeurs de même type. La fonction c() (combine) crée un vecteur en combinant des valeurs. Par exemple, la syntaxe c(2, 4, 6) renvoie un vecteur constitué de ces trois valeurs : ce vecteur a une taille (3), ses valeurs peuvent être utilisées dans d'autres calculs, ses valeurs ont un ordre par lequel elles peuvent être désignées (la 2e valeur est 4).
La plupart des objets utilisés dans R peuvent être décomposés en un ensemble de vecteurs. Le tableau suivant (cf. Figure 2.1.2) est constitué de 5 lignes (individus) et 3 colonnes (variables) : il peut être considéré comme la combinaison de trois vecteurs composés de cinq valeurs ou comme la combinaison de cinq vecteurs composés de trois valeurs. Dans le premier cas, le vecteur stocke une variable ; dans le second cas, le vecteur stocke un individu.
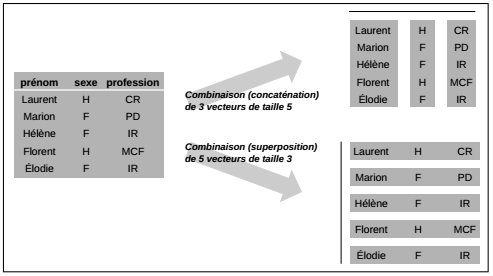
Lorsqu'on crée un tableau, qu'on le remplit ou qu'on en extrait des informations, il est utile de garder à l'esprit qu'il est décomposable en un ensemble de vecteurs.
Le facteur (factor) est un vecteur un peu particulier qu'il est nécessaire de savoir manipuler. Il arrive souvent de transformer des facteurs en vecteurs et des vecteurs en facteurs. Il est aussi fréquent d'empêcher la création de facteurs à la création ou l'importation de tableaux. En effet, dans l'importation d'un tableau externe, les champs alphanumériques sont automatiquement transformés en facteurs, sauf à spécifier le contraire avec l'argument stringsAsFactors = FALSE.
Le facteur est très pratique pour stocker de véritables variables qualitatives, c'est-à-dire des variables dont les modalités caractérisent des sous-populations : pour des individus, le sexe ou la catégorie socioprofessionnelle; pour des communes, le département d'appartenance ou la couleur politique. Ce format permet d'éviter des erreurs de codage et il est particulièrement intéressant pour les catégories aux intitulés très longs. Par exemple, pour travailler sur des catégories socioprofessionnelles, il est plus pertinent de coder ces catégories avec des entiers auxquels sont associées des étiquettes plutôt que de coder directement les longs intitulés de ces catégories (« Cadres et professions intellectuelles supérieures », etc.). Le format facteur fait aussi gagner de la mémoire dans un tel cas puisque ces longues étiquettes ne sont pas répétées autant de fois qu'il y a d'individus.
En revanche, le facteur n'est pas adapté lorsqu'il s'agit de stocker des identifiants : le code d'identification des individus ou le code Insee des communes par exemple. Par définition, un identifiant est un champ qui prend une valeur unique pour chaque individu et permet ainsi de l'identifier. Le format facteur ne fournit dans ce cas aucun gain de mémoire puisqu'il y a autant d'étiquettes distinctes que d'individus. Plus important, le facteur est constitué de niveaux et d'étiquettes et cette combinaison est 16 Prise en main et manipulation des données particulièrement dangereuse lors des manipulations de tableaux qui impliquent leur identifiant. Toute jointure de tableaux sur des identifiants stockés en format facteur est à proscrire.
2-1-3. Déclarer des objets avec l'opérateur d'assignation▲
Pour créer et initialiser un objet, on utilise l'opérateur d'assignation <-. Sans l'opérateur d'assignation le résultat de l'instruction est renvoyé dans la console mais aucun objet n'est créé. La syntaxe générale est la suivante “Mon objet <- Contenu de mon objet”.
2.
3.
4.
5.
6.
7.
8.
1 + 1 # renvoie le résultat dans la console
## [1] 2
myObject <- 1 + 1 # assigne le résultat à un objet
myObject # affiche le contenu de l'objet
## [1] 2
Dans RStudio, chaque objet créé est affiché dans la fenêtre Environment accompagné de certaines de ses caractéristiques. Dans cette fenêtre, on peut cliquer sur les objets créés pour les visualiser.
Il y a deux méthodes pour déclarer des objets :
- de façon implicite, c'est-à-dire en assignant à l'objet créé le résultat d'une fonction qui renvoie par défaut un certain type d'objet ;
- de façon explicite, c'est-à-dire en déclarant le type de l'objet au moment de sa création.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
# déclaration implicite
myObject <- 1 + 1
class(myObject)
## [1] "numeric"
# déclaration explicite
myFactor <- factor(c(1, 1, 2, 1, 2, 2),
labels = c("Homme", "Femme"))
class(myFactor)
## [1] "factor"
On peut créer les différents types d'objets cités précédemment avec des fonctions éponymes : vector(), matrix(), list(), data.frame(), etc. Dans les trois exemples qui suivent, on utilise la fonction c() pour créer un vecteur simple (Créer un vecteur); pour créer un vecteur et le mettre en forme dans une matrice à 3 lignes et 4 colonnes (Créer une matrice); pour créer un vecteur de deux noms (ID et CLASS), qu'on assigne comme noms de colonnes à un data.frame (Créer un data.frame).
Créer un vecteur :
La fonction c() renvoie un vecteur en combinant des valeurs d'un certain type, par exemple de valeurs numériques (numeric) ou des valeurs alphanumériques (character).
2.
3.
4.
5.
6.
7.
8.
9.
vecNum <- c(1, 2, 3, 4, 5)
vecNum
## [1] 1 2 3 4 5
vecChar <- c("CP", "CE1", "CE2", "CM1", "CM2")
vecChar
## [1] "CP" "CE1" "CE2" "CM1" "CM2"
Créer une matrice :
La création d'une matrice se fait à partir d'un vecteur en explicitant le nombre de lignes et de colonnes à construire. Par défaut R remplit d'abord les colonnes de haut en bas puis de gauche à droite comme l'illustre l'exemple ci-dessous.
2.
3.
4.
5.
6.
7.
matNum <- matrix(c(1: 12), nrow = 3, ncol = 4)
matNum
## [,1] [,2] [,3] [,4]
## [1,] 1 4 7 10
## [2,] 2 5 8 11
## [3,] 3 6 9 12
Créer un data.frame :
Pour créer un objet data.frame on a recours à la fonction éponyme qui peut prendre en entrée une matrice, ou une série de vecteurs. Le dernier argument stringAsFactors = FALSE empêche la transformation automatique des vecteurs de chaînes de caractères en facteurs. Pour modifier ou afficher les noms de colonnes, sous la forme d'un vecteur alphanumérique, on peut utiliser la fonction names() ou colnames(). Les noms de colonnes peuvent être modifiés a posteriori (Option 1) ou dès la déclaration de l'objet (Option 2).
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
# Option 1
dfNumChar <- data.frame(vecNum,
vecChar,
stringsAsFactors=FALSE)
colnames(dfNumChar)
## [1] "vecNum" "vecChar"
colnames(dfNumChar) <- c("ID", "CLASS")
# Option 2
dfNumChar <- data.frame(ID = vecNum,
CLASS = vecChar,
stringsAsFactors = FALSE)
dfNumChar
## ID CLASS
## 1 1 CP
## 2 2 CE1
## 3 3 CE2
## 4 4 CM1
## 5 5 CM2
2-1-4. Modifier le type d'un objet▲
Il est possible de transformer un objet d'un certain type en un autre type, avec des fonctions suivant la syntaxe suivante as. suivi du type choisi, par exemple as.matrix, as.vector, etc. Ces transformations peuvent être classés en trois catégories : les transformations sans perte d'information, les transformations avec perte d'information, les transformations détruisant la structure de l'objet.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
dfNum <- data.frame(col1 = vecNum,
col2 = c(2, 5, 8, 7, 8),
stringsAsFactors = FALSE)
# transformation sans perte
matNum <- as.matrix(dfNum)
# transformation avec perte des étiquettes
myVec <- as.numeric(myFactor)
myVec
## [1] 1 1 2 1 2 2
# perte d'une dimension, perte des noms des champs
vecFromMat <- as.vector(matNum)
vecFromMat
## [1] 1 2 3 4 5 2 5 8 7 8
2-1-5. Désigner des lignes, des colonnes ou des valeurs▲
L'un des gros avantages de R vis-à-vis des logiciels de statistiques classiques est qu'il permet de désigner très facilement un ensemble de valeurs contenues dans un objet, comme dans un tableur où on peut désigner une cellule en précisant sa ligne et sa colonne. La façon de désigner ces éléments diffère selon le type d'objet dans lequel ils sont stockés.
Pour les vecteurs et les matrices, on fait appel à l'index souhaité entre crochets ([]) et on désigne la dimension de l'objet souhaitée en les séparant par des virgules : une dimension pour un vecteur, deux pour une
matrice, trois pour un cube, etc. Par exemple, on appelle la deuxième valeur d'un vecteur en écrivant monVecteur[2].
Dans le cas des tableaux (data.frame) et des listes, les crochets s'utilisent de la même façon que dans le cas précédent, mais il est aussi possible de désigner l'élément souhaité précédé d'un dollar ($). Les utilisateurs habitués aux tableaux avec des intitulés de colonne accèderont de cette façon à la variable de leur choix : MonTableau$VARIABLE. Pour créer une nouvelle variable, il suffit d'assigner des valeurs à une variable dont le nom n'existe pas encore dans le tableau. Si l'utilisateur assigne des valeurs à une variable dont le nom existe déjà dans le tableau, les valeurs originelles seront écrasées par les nouvelles valeurs.
En laissant l'indexation vide pour une dimension donnée, on la récupère dans son ensemble. Par exemple, pour un tableau à deux dimensions (data.frame ou matrix), l'instruction monTableau[2, 3] renverra la valeur située à la deuxième ligne et troisième colonne alors que l'instruction monTableau[2, ] renverra la deuxième ligne et l'ensemble des colonnes. Testez les lignes suivantes :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
# Sélection
vecNum[ 2]
matNum[ 3, 2]
matNum[, 2]
dfNumChar[, 2]
dfNumChar$CLASS
dfNumChar$CLASS[ 2]
dfNumChar[ 3, 2]
# Sélection et assignation
vecCharLong <- vecChar
vecCharLong[ 1] <- "Cours primaire"
vecCharLong[ 2: 3] <- "Cours élémentaire"
vecCharLong[ 4: 5] <- "Cours moyen"
dfNumChar$CLASS <- "CP"
Les utilisateurs ayant une culture de tableur ou de logiciel de statistique classique utilisent fréquemment des tableaux de variables. Ils trouveront pesant de sans cesse devoir faire référence au tableau accompagné du $ avant de désigner la variable (monTableau$VARIABLE). Il est possible d'attacher le tableau et de désigner directement les variables par leur nom avec la fonction attach() (et detach() pour le détacher). Cependant, ces fonctions ne permettent pas de travailler directement sur l'objet data.frame mais sur une copie de celui-ci. Pour cette raison elles représentent une source d'erreur et leur usage est fortement déconseillé. La fonction with() peut être utilisée dans le même but et elle est sans danger.
2.
3.
4.
5.
6.
7.
mean(dfNum$col2)
## [1] 6
with(dfNum, mean(col2))
## [1] 6
2-1-6. Traitement des valeurs manquantes▲
Dans R, les valeurs manquantes sont notées NA (not available). Par défaut, la plupart des fonctions de base (somme, moyenne, minimum, maximum, etc.) n'acceptent pas les variables contenant des valeurs manquantes, ce qui peut être déroutant pour les utilisateurs d'autres logiciels, en particulier les tableurs. Dans ce cas, l'utilisateur doit préciser l'action à réaliser avec les valeurs manquantes, la plus simple étant souvent de les supprimer (remove) : na.rm=TRUE.
2.
3.
4.
5.
6.
7.
8.
vecTest <- c(2, 4, NA, 6)
mean(vecTest)
## [1] NA
mean(vecTest, na.rm = TRUE)
## [1] 4
La fonction is.na() est utile pour se renseigner sur la présence de valeurs manquantes, elle renvoie un vecteur booléen indiquant la position de ces valeurs manquantes. Cet index des valeurs manquantes peut être réutilisé de plusieurs façons : combiné avec la fonction any(), il permet de savoir s'il y a une ou plusieurs valeurs manquantes dans une variable ; intégré avec un assignement il peut servir à recoder ces valeurs manquantes.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
# Index des valeurs manquantes
is.na(vecTest)
## [1] FALSE FALSE TRUE FALSE
# Présence de valeurs manquantes dans la variable
any(is.na(vecTest))
## [1] TRUE
# Option équivalente à la ligne précédente
anyNA(vecTest)
## [1] TRUE
# Recodage des valeurs manquantes en 0
vecTest[ is.na(vecTest)] <- 0
2-1-7. Se renseigner sur les objets et leur contenu▲
Il est fondamental de toujours savoir quels objets ont étés créés durant la session, sur quels types d'objets on travaille, quelle est leur structure, leur taille ou leur contenu. Les commandes suivantes sont donc indispensables pour éviter de travailler à l'aveugle.
Lister et effacer les objets déclarés dans la session :
2.
3.
ls() # lister les objets
rm(vecNum) # effacer un seul objet
rm(list = ls()) # effacer tous les objets
Se renseigner sur la nature et le contenu des objets :
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
21.
22.
data(cars)
str(cars) # structure de l'objet
## 'data.frame': 50 obs. of 2 variables:
## $ speed: num 4 4 7 7 8 9 10 10 10 11 ...
## $ dist : num 2 10 4 22 16 10 18 26 34 17 ...
class(cars) # type de l'objet
## [1] "data.frame"
dim(cars) # dimension de l'objet
## [1] 50 2
is.matrix(cars) # test sur le type de l'objet
## [1] FALSE
is.data.frame(cars) # test sur le type de l'objet
## [1] TRUE
Dans l'exemple ci-dessus, le jeu de données utilisé (cars) est un jeu de données contenu dans la base R, il est chargé avec la fonction data(). Pour obtenir la liste de tous les jeux de données d'exemple disponibles, on peut écrire la fonction data() puis utiliser la touche Tab pour l'autocomplétion.
2-2. Importation et exportation▲
Cette section permet d'introduire les commandes nécessaires à l'importation et l'exportation de données externes dans les formats les plus couramment utilisés.
2-2-1. Le répertoire de travail▲
Avant d'importer et d'exporter des données, il est nécessaire de savoir
se renseigner sur le répertoire de travail (working directory) et éventuellement le modifier. Deux fonctions simples permettent de gérer ce répertoire :
getwd() : renvoie le chemin du working directory ;setwd("/chemin/dossier") : spécifie le working directory.
Certaines fonctionnalités introduites par RStudio rendent plus facile la gestion de ce répertoire. Dans le menu Session, plusieurs boutons permettent de fixer rapidement le répertoire de travail. En outre, RStudio permet d'organiser le travail en projets grâce à l'onglet Projects, situé en haut à droite de l'interface. Utiliser ces fonctionnalités facilite l'accès à des espaces de travail différenciés correspondant aux différents travaux en cours de l'utilisateur.
2-2-2. Les for mats de données▲
Les objets peuvent être créés directement sous R ou importés depuis des bases de données externes. Plusieurs formats sont disponibles :
- formats texte (csv, txt) (importation directe avec r-base),
- formats tableurs (xls, ods) (l'importation sous R nécessite des packages spécifiques),
- formats spécifiques des logiciels de statistiques (sas7bdat pour SAS, sav pour SPSS, etc.),
- formats spécifiques de bases de données (SQLite, MySQL, etc.).
2-2-3. Codage des caractères et des séparateurs▲
Il faut bien distinguer le format d'un fichier, indiqué par son extension (xls, csv, etc.), du codage (encoding) de ses caractères. Il est toujours utile de savoir quel est le codage des documents qu'on utilise, en général UTF8 ou Latin1. Il arrive souvent qu'on importe un fichier avec des champs de caractères en français et que les accents et les apostrophes soient mal affichés. Ces problèmes surviennent en général dans le cas d'une utilisation de plusieurs systèmes d'exploitation (Linux-Windows par exemple), ou quand les données sont manipulées sur des ordinateurs ayant des normes linguistiques différentes.
Un autre aspect des conventions d'écriture doit être souligné, celui du codage des séparateurs de champs et des séparateurs décimaux. Un fichier de données au format texte (txt ou csv) suivant les conventions anglosaxonnes aura pour séparateur de champs la virgule et pour séparateur décimal le point. Un fichier aux conventions françaises aura pour séparateur de champs le point-virgule et pour séparateur décimal la virgule. Ces informations seront nécessaires lors de l'importation du fichier.
2-2-4. Importer des données en format texte▲
Les données stockées dans des formats de tableurs (xls, ods) peuvent être importées directement, mais on évite bien des soucis en les enregistrant au préalable sous un format texte (txt ou csv). Il y a ensuite deux façons d'importer dans RStudio un fichier de ce type :
- par l'interface graphique : dans RStudio, dans l'onglet Environment de la fenêtre placée en haut à droite de l'écran, on utilise le bouton Import Dataset. On peut visualiser les données brutes et les données telles qu'elles apparaitront après importation. Cela permet de modifier si nécessaire le séparateur de champs, le séparateur décimal et d'inclure ou non l'intitulé de colonne ;
- en ligne de commande : on utilise la fonction read.table() en spécifiant le chemin et le nom du fichier, le type de séparateur de champs (sep), le type de séparateur décimal (dec), le fait d'inclure ou non les intitulés de colonnes (header). Le codage du fichier d'origine peut aussi être précisé : si le codage du système est UTF-8 et que le fichier importé affiche mal les accents et les apostrophes, l'argument de codage (encoding) peut être utilisé (latin1 en général).
2-2-5. Exporter des données en format texte▲
Pour exporter un tableau de données en format texte on utilise la fonction write.table() en spécifiant, comme pour la fonction read.table(), le nom et le chemin du fichier à créer, les séparateurs, etc. Ces fonctions peuvent être remplacées par des fonctions qui présentent certains réglages par défaut, en particulier des séparateurs, read.csv() par exemple.
2-2-6. Importation et exportation depuis/vers d'autres for mats▲
Le package foreign permet de lire et d'écrire des fichiers dans les formats des grands logiciels commerciaux. Il comprend les fonctions read.spss() pour les formats sav, read.xport() pour le format xport de SAS. Pour SAS il existe aussi le package sas7bdat qui permet d'importer directement des fichiers dans ce format.
Il est également possible de stocker des données dans une base de données externes, PostgreSQL, SQLite ou autre et d'y accéder depuis R avec les packages correspondants (RPostgreSQL, RSQLite, etc.).
2-3. Recoder et trier▲
Les sélections et les recodages se font principalement avec les opérateurs logiques suivants :
- x est égal à y : x == y
- x n'est pas égal à y : x != y
- x est strictement supérieur à y : x > y
- x est strictement inférieur à y : x < y
- x est supérieur ou égal à y : x >= y
- x est inférieur ou égal à y : x <= y
- A ∩ B (opérateur ET, intersection de deux conditions) : A & B
- A ∪ B (opérateur OU, union de deux conditions) : A | B
2-3-1. Sélectionner et recoder▲
Le réflexe des utilisateurs de logiciels de statistiques classiques est de vouloir faire des sélections et des recodages avec des opérateurs conditionnels (if [...] then). Par exemple on a une liste d'individus dont l'âge est renseigné, et on souhaite discrétiser cette variable en deux classes 28 Prise en main et manipulation des données d'âge selon que les individus sont mineurs ou majeurs. Une pseudosyntaxe classique donnerait quelque chose comme :
tant que (ligne individu != 0)
{if AGE < 18 then CLASS = Min else CLASS = Maj}À partir des méthodes présentées jusqu'ici, le premier réflexe serait d'utiliser l'indexation des valeurs : pour chaque numéro de ligne où la variable AGE est inférieure à 18, écrire « Min » dans la variable CLASS ; pour chaque numéro de ligne où la variable AGE est supérieure ou égale à 18, écrire « Maj » dans la variable CLASS.
2.
3.
4.
5.
6.
7.
8.
9.
10.
dfIndiv <- data.frame(ID = c(1, 2, 3, 4, 5),
AGE = c(12, 17, 24, 45, 8),
SEX = c("H", "H", "F", "F", "F"))
# Assignation conditionnelle dans une nouvelle variable
dfIndiv$CLASS[dfIndiv$AGE < 18] <- "Min"
dfIndiv$CLASS[dfIndiv$AGE >= 18] <- "Maj"
dfIndiv$CLASS
## [1] "Min" "Min" "Maj" "Maj" "Min"
La fonction ifelse() simplifie ce travail. Elle sert à assigner une valeur à une variable en fonction d'un test conditionnel et peut être appliquée sur un tableau. Cette fonction s'utilise de la façon suivante :
ifelse(test, valeur si vrai, valeur si faux)2.
3.
4.
dfIndiv$CLASS <- ifelse(dfIndiv$AGE < 18, "Min", "Maj")
dfIndiv$CLASS
## [1] "Min" "Min" "Maj" "Maj" "Min"
Attention, il ne faut pas confondre la fonction ifelse() avec la fonction if() qui sert à exécuter une instruction en fonction du résultat du test (cf. Section 3.2La structure conditionnelle if…else).
La fonction subset() est très utile pour sélectionner des parties d'un tableau en fonction de la valeur de certaines variables ou pour conserver certaines colonnes d'un tableau. Cette fonction, comme la fonction ifelse(), peut prendre comme argument un test à conditions multiples utilisant les opérateurs de comparaison.
2.
3.
4.
5.
subset(dfIndiv, dfIndiv$SEX == "F" & dfIndiv$AGE > 18)
## ID AGE SEX CLASS
## 3 3 24 F Maj
## 4 4 45 F Maj
Le package plyr (puis dplyr, cf. Section 2.5Packages polyvalents) propose deux fonctions utiles pour le recodage : mapvalues() et revalue() qui permettent d'assigner de nouvelles valeurs à un vecteur numérique, alphanumérique ou à un facteur.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
library(plyr)
revalue(dfIndiv$SEX, c("F" = "Femme", "H" = "Homme"))
## [1] Homme Homme Femme Femme Femme
## Levels: Femme Homme
mapvalues(dfIndiv$SEX,
from = c("F", "H"),
to = c("Femme", "Homme"))
## [1] Homme Homme Femme Femme Femme
## Levels: Femme Homme
Une opération de recodage très fréquente est la discrétisation, qui consiste à découper une variable continue en classes. Il serait bien sûr possible de faire ce recodage avec une suite de tests conditionnels définis dans un emboîtements de fonctions ifelse(). Cependant, R dispose d'une fonction plus pratique pour discrétiser une variable continue : la fonction cut().
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
valBreaks <- c(8, 18, 45)
dfIndiv$AGEDISCRET <- cut(dfIndiv$AGE,
breaks = valBreaks,
include.lowest = TRUE,
right = FALSE)
dfIndiv
## ID AGE SEX CLASS AGEDISCRET
## 1 1 12 H Min [8,18)
## 2 2 17 H Min [8,18)
## 3 3 24 F Maj [18,45]
## 4 4 45 F Maj [18,45]
## 5 5 8 F Min [8,18)
Cette fonction prend un vecteur numérique comme argument et le découpe en fonction de seuils (breaks) définis au préalable. Les intervalles peuvent être fermés sur la droite ou sur la gauche (right) et inclure la valeur maximum ou minimum selon le cas (include.lowest). Le vecteurs de seuils est ici défini à la main, mais il est bien sûr possible d'utiliser des fonctions pour définir des seuils fréquemment utilisés, comme les quantiles avec la fonction quantile() Cette manipulation est présentée dans la Section 3.3.2Discrétisation automatique.
2-3-2. Trier▲
Pour trier les valeurs d'un objet dans un certain ordre, croissant, décroissant ou alphabétique, deux fonctions peuvent être utilisées : sort() et order(). Il y a deux différences majeures entre ces fonctions : sort() ne permet de trier les valeurs que d'une seule variable, alors que order() permet de trier un tableau de valeurs en fonction d'une ou plusieurs variables. Autre différence notable, order() renvoie le rang tenu par les valeurs, alors que sort() renvoie les valeurs elles-mêmes.
Ainsi pour trier l'ensemble d'un tableau selon les valeurs prises par une variable, il faudra toujours utiliser order(). L'intérêt de la fonction order() réside dans la possibilité de trier un tableau de plusieurs variables selon l'ordre des valeurs prises par une variable en particulier. Si data est le nom d'un tableau et data$var le nom de la variable selon laquelle le tableau doit être trié, on utilise l'instruction data[order(data$var), ]. Cette syntaxe indique qu'il faut ordonner les lignes du tableau (première dimension entre crochets) en fonction du rang des valeurs de la variable var.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
# Trier un vecteur
sortedAge <- sort(dfIndiv$AGE)
sortedAge
## [1] 8 12 17 24 45
# Trier un tableau selon une variable
sortedIndiv <- dfIndiv[ order(dfIndiv$AGE), ]
# Trier un tableau selon plusieurs variables
sortedCars <- cars[ order(cars$speed, cars$dist), ]
La syntaxe de la fonction order() est peu lisible. Le package plyr (puis dplyr, cf. Section 2.5Packages polyvalents) propose une fonction plus pratique pour trier un tableau de données : arrange().
library(plyr)
sortedCars <- arrange(cars, speed, dist)Si trier les valeurs d'un vecteur numérique ne pose pas de problème particulier, il est plus délicat de trier un facteur. En effet, celui-ci est composé de valeurs (levels) et d'étiquettes (labels), le tri peut s'appliquer à l'un ou l'autre selon la syntaxe employée.
2.
3.
4.
5.
6.
7.
8.
sexFactor <- factor(c(1, 1, 2, 1, 2, 2), labels = c("H", "F"))
as.numeric(sexFactor)
## [1] 1 1 2 1 2 2
as.character(sexFactor)
## [1] "H" "H" "F" "H" "F" "F"
Si le tri s'applique aux valeurs, les hommes (codés 1) seront placés avant les femmes (codées 2). Si le tri s'applique aux étiquettes, les femmes (lettre f) seront placées avant les hommes (lettre h). Il est donc plus sûr de transformer le facteur avec les fonctions as.character() ou as.numeric() puis d'appliquer explicitement le tri aux étiquettes ou aux valeurs.
2-4. Manipulation avancée▲
La première partie de ce chapitre a présenté des procédures simples de calcul, de sélection, de recodage et de tri. La section suivante introduit les principales techniques de manipulation avancée des données, à savoir les techniques qui permettent de transformer la structure même des données par des agrégations, des jointures et des transpositions.
Les données utilisées ici concernent les communes de Paris et de la petite couronne, c'est-à-dire les départements 75, 92, 93 et 94. L'importation se fait en utilisant les fonctions présentées plus haut (cf. Section 2.2Importation et exportation) : read.table(), read.csv() ou interface graphique de RStudio.
2.
3.
4.
5.
6.
socEco9907 <- read.csv("data/SocEco9907.csv",
sep = ";",
stringsAsFactors = FALSE)
popCom3608 <- read.csv("data/PopCom3608.csv",
sep = ";",
stringsAsFactors = FALSE)
2-4-1. Superposition, concaténation, jointure▲
De la même façon que la fonction c() combine des valeurs dans un vecteur ou une liste, les fonctions rbind() et cbind() permettent de prendre un vecteur, une matrice ou un tableau et de les combiner par ligne ou par colonne. La combinaison par ligne (row) est appelée superposition (rbind) et la combinaison par colonne (column) est appelée concaténation (cbind).
Ces deux fonctions doivent être utilisées avec précaution. La fonction rbind() permet de superposer deux tableaux dont les colonnes sont identiques, c'est-à-dire qui contiennent le même nombre de colonnes et dont les noms sont les mêmes. Prenant en compte le nom de colonne, la fonction permet une superposition même si les colonnes ne sont pas dans le même ordre. Si les colonnes sont différentes, la fonction renvoie un message d'erreur.
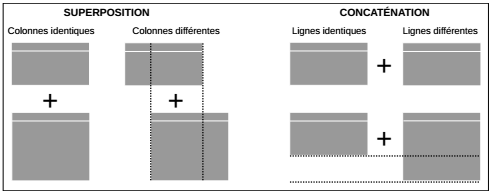
De la même façon, si les deux tableaux ont un nombre de lignes différent, la fonction cbind() renvoie un message d'erreur. En revanche, si le nombre de lignes est le même, la fonction concatène les deux tableaux sans se soucier de leur contenu. Elle est donc inadaptée pour combiner des données de différentes sources sur les mêmes individus statistiques (individus, communes, régions, etc.). Il faut dans ce cas faire une concaténation selon un identifiant qui garantit que l'identité de chaque individu est conservée. Cette concaténation avec identifiant est appelée jointure.
C'est la fonction merge() qui permet d'effectuer cette jointure de deux tableaux en fonction d'une variable commune. L'exemple suivant joint les deux tableaux socEco9907 et popCom3608 à l'aide de l'identifiant commun nommé CODGEO dans les deux tableaux.
jointCom <- merge(socEco9907, popCom3608, by = "CODGEO")Le cas présenté ici est le plus simple : les deux tableaux ont exactement les mêmes individus statistiques (i.e. les lignes se correspondent deux à deux) et le nom de la variable identifiant (code de la commune) est le même dans les deux tableaux.
La fonction merge() s'adapte à tous les cas de jointures grâce à des arguments complémentaires. L'argument by peut être divisé en by.x et by.y pour désigner le nom de la colonne qui sert d'identifiant dans chacun des deux tableaux. Les arguments all, all.x, all.y servent dans les cas où les deux tableaux n'ont pas les mêmes individus statistiques. Quatre grands cas de jointures peuvent être distingués :
- inner join : conservation des seules lignes communes aux deux tableaux (
all=FALSE) ; - left join : conservation de toutes les lignes du tableau de gauche même si elles n'ont pas de lignes correspondantes dans le tableau de droite (all.x
=TRUE) ; - right join : conservation de toutes les lignes du tableau de droite même si elles n'ont pas de lignes correspondantes dans le tableau de gauche (all.y
=TRUE) ; - outer join : conservation de toutes les lignes des deux tableaux (
all=TRUE).
2-4-2. Agrégations et traitements par blocs▲
Les procédures d'agrégation servent en premier lieu à calculer un résumé numérique selon une variable d'agrégation. Cette procédure met en jeu trois arguments : la variable sur laquelle est calculé le résumé numérique (variable quantitative), la variable d'agrégation (variable qualitative) et la fonction d'agrégation (somme, moyenne, max, etc.). Trois fonctions sont utiles dans ce cas : by(), tapply() et aggregate().
L'exemple suivant consiste à calculer la population totale par département à partir de la variable population renseignée à la commune. Il demande d'abord de créer une variable indiquant le département par extraction des deux premiers caractères du code de la commune. Cette variable est créée avec la fonction substr() (substring) qui prend comme arguments la variable alphanumérique (le code Insee) et la position du premier et du dernier caractère à extraire. La fonction tapply() est employée car c'est la plus simple (et la plus limitée) des trois : elle n'accepte qu'une seule variable de calcul et qu'une seule variable d'agrégation.
2.
3.
4.
5.
6.
7.
8.
9.
10.
popCom3608$DEP <- substr(popCom3608$CODGEO, 1, 2)
tapply(popCom3608$POP2008, popCom3608$DEP, sum)
## 75 92 93 94
## 2211297 1549619 1506466 1310876
tapply(popCom3608$POP1936, popCom3608$DEP, sum)
## 75 92 93 94
## 2829755 1019627 776378 685204
Il est souvent utile de faire ce type de traitement sur plusieurs variables quantitatives et/ou plusieurs variables d'agrégation. C'est dans ce cas que les fonctions by() et aggregate() sont employées. Cette procédure peut servir à renvoyer un simple résultat à analyser mais également à recréer une information à un niveau spatial plus agrégé. À partir des données communales, il serait par exemple intéressant de créer des tableaux au niveau départemental regroupant les mêmes variables de population et d'emploi.
2.
3.
4.
5.
6.
7.
popDep <- aggregate(popCom3608[ , c("POP1936", "POP2008")],
by = list(popCom3608$DEP),
FUN = sum)
popDep$EVOL3608 <- with(popDep, POP2008 / POP1936 - 1)
popDep$EVOL3608
## [1] -0.2186 0.5198 0.9404 0.9131
On constate ici le contraste entre la décroissance de la population au centre de l'agglomération parisienne (-0,22 % pour le département 75) et la croissance de la population des départements de petite couronne (92, 93, 94) entre 1936 et 2008.
Au-delà du simple calcul de résumés numériques selon une ou plusieurs variables d'agrégation, il est parfois nécessaire d'appliquer des fonctions plus compliquées à un tableau de données divisé en blocs. Pour ce type de traitement (split-apply-combine strategy), il faudra utiliser les fonctions du package plyr. Elles fonctionnent comme celles qui viennent d'être présentées avec les trois mêmes arguments principaux (données, indices, fonction). Les deux premières lettres de leur nom indique le type des objets en entrée et en sortie de la fonction, par exemple ddply() prend en entrée un tableau (data.frame) et renvoie un tableau. Plusieurs options sont possibles, par exemple dlply() (tableau, liste) ou mdply() (matrice, tableau).
Le traitement par blocs peut être combiné avec tout type de fonctions, que celles-ci soient propres à l'utilisateur ou implémentées dans les packages existants. Parmi ces dernières, trois sont spécialement utiles : transform(), summarise() et subset(). Ce premier exemple calcule la moyenne et l'écart-type de la population communale de 2008 par département.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
depAgg08 <- ddply(.data = popCom3608,
.variables = "DEP",
.fun = summarise,
POPMEAN08 = mean(POP2008),
POPSD08 = sd(POP2008))
str(depAgg08)
## 'data.frame': 4 obs. of 3 variables:
## $ DEP : chr "75" "92" "93" "94"
## $ POPMEAN08: num 110565 43045 37662 27891
## $ POPSD08 : num 71227 26127 24250 21623
Ce second exemple standardise les populations communales en 2008 pour chacun des quatre départements considérés. Ces variables centrées-réduites (moyenne = 0, écart-type = 1) sont calculées avec la fonction scale() qui est appliquée à chaque sous-ensemble correspondant à chacun des quatre départements.
2.
3.
4.
standPop08 <- ddply(.data = popCom3608,
.variables = "DEP",
.fun = transform,
STDPOP08 = scale(POP2008))
Le traitement par blocs est particulièrement utile pour analyser des données géographiques puisqu'il est fréquent de faire des traitements à plusieurs niveaux emboîtés : communes, départements, régions par exemple.
2-4-3. Transposition variables-observations▲
Il est courant d'avoir à transformer la structure même des données en réorganisant les lignes et les colonnes. Cette procédure, nommée ici transposition variables-observations, se fait avec les fonctions du package reshape2. Elle ne doit pas être confondue avec la transposition du calcul matriciel qui consiste à inverser les lignes et les colonnes et qui se fait avec la fonction t().
Lorsqu'on travaille sur des tableaux statistiques, une ligne ne représente pas toujours un individu statistique et une colonne une variable. Dans certains cas, ce type de formalisation n'est pas pertinent ou n'existe pas. Par exemple dans une matrice origine-destination (origine des flux en ligne, destination des flux en colonne) la distinction entre individu et variable n'a pas de sens.
De façon générale, on distingue deux types de format de données : d'une part, le format large (wide), qui est celui utilisé jusqu'à présent dans le manuel et dans lequel une ligne représente un individu statistique caractérisés par plusieurs variables en colonne. D'autre part, le format long (long), qui est très utilisé par exemple dans les bases de données temporelles. Pour manipuler des données longitudinales (la même variable observée à plusieurs pas de temps), il y a toujours deux options de stockage :
- sous forme wide, une ligne représente un individu statistique et chaque colonne la mesure pour une date donnée ;
- sous forme long, il y a trois colonnes : l'identifiant de l'individu, la date de la mesure et la valeur de la mesure. Ainsi chaque ligne représente une combinaison unique individu-date.
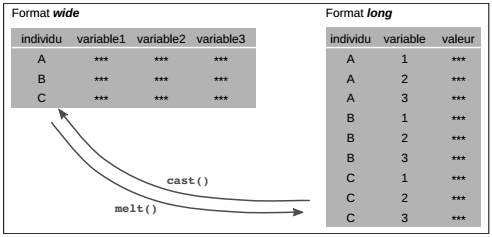
Les deux formats représentent bien sûr exactement la même information et c'est dans la facilité d'usage selon différents objectifs que réside le choix du format. Ce sont les fonctions melt() et cast() du package reshape2 qui permettent le passage de l'un à l'autre.
Ce package part d'une distinction essentielle des variables d'un tableau entre variables identifiants et des variables de mesure. Les variables identifiants (id variables) sont discrètes, elles désignent les unités d'enregistrement. Les variables de mesures (measured variables) enregistrent les mesures correspondant à chaque unité d'enregistrement. Il est conseillé, mais non nécessaire, de commencer la transposition par la fonction melt(), pour bien identifier les variables identifiants et les variables de mesure. Cette fonction permet de préparer les données en vue d'une utilisation avec d'autres commandes offertes par le même package.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
library(reshape2)
meltedPopCom <- melt(popCom3608,
id.vars = c("CODGEO", "LIBELLE", "SURF"),
variable.name = "YEAR")
head(meltedPopCom)
## CODGEO LIBELLE SURF YEAR value
## 1 75101 Paris 1er Arrondissement 183 POP1936 37062
## 2 75102 Paris 2e Arrondissement 99 POP1936 41445
## 3 75103 Paris 3e Arrondissement 117 POP1936 63571
## 4 75104 Paris 4e Arrondissement 160 POP1936 62547
## 5 75105 Paris 5e Arrondissement 254 POP1936 97396
## 6 75106 Paris 6e Arrondissement 215 POP1936 81403
Dans l'exemple précédent, l'argument id.vars sert à désigner les variables identifiants, l'argument variable.name sert à nommer la variable qui va stocker les mesures et l'argument measure.vars n'est pas précisé. N'étant pas précisé, la fonction melt() comprend que toutes les variables non désignées comme identifiant sont des variables de mesure.
La fonction cast() se décline suivant le type d'objet renvoyé : acast() pour une matrice (array) et dcast() pour un tableau (data.frame). La formule utilisée par la fonction cast() sépare les variables à mettre en colonne des variables à mettre en ligne avec le tilde (symbole ˜ qui s'écrit avec AltGr + 2 sur un clavier français). Elle accepte plusieurs variables à mettre en colonne et/ou plusieurs variables à mettre en ligne, en ce cas la formule utilise l'opérateur + :
column_var ˜ row_var
cvar_1 + cvar_2 + cvar_n ˜ rvar_1 + rvar_2 + rvar_nVoici un exemple de transposition variables-observations permettant de revenir au tableau initial wide, avant son re-positionnement au format long par la fonction melt().
2.
3.
4.
5.
6.
7.
8.
9.
10.
castedByCode <- dcast(meltedPopCom, CODGEO ~ YEAR)
head(castedByCode[, 0: 6])
## CODGEO POP1936 POP1954 POP1962 POP1968 POP1975
## 1 75101 37062 37330 36543 32332 22793
## 2 75102 41445 41744 40864 35357 26328
## 3 75103 63571 64030 62680 56252 41706
## 4 75104 62547 62998 61670 54029 40466
## 5 75105 97396 98099 96031 83721 67668
## 6 75106 81403 81991 80262 70891 56331
La transposition permet aussi de récupérer, à partir du format long, le code géographique en colonne et le temps en ligne :
2.
3.
4.
5.
6.
7.
8.
9.
10.
castedByYear <- dcast(meltedPopCom, YEAR ~ CODGEO)
head(castedByYear[, 0: 6])
## YEAR 75101 75102 75103 75104 75105
## 1 POP1936 37062 41445 63571 62547 97396
## 2 POP1954 37330 41744 64030 62998 98099
## 3 POP1962 36543 40864 62680 61670 96031
## 4 POP1968 32332 35357 56252 54029 83721
## 5 POP1975 22793 26328 41706 40466 67668
## 6 POP1982 18509 21203 36094 33990 62173
Le passage d'un format à l'autre est fréquent dès qu'il s'agit de traiter des flux entre des lieux. Une information de ce type comprend un couple de lieux (origine-destination) et des mesures correspondantes à ce couple : une distance ou un flux par exemple. L'exemple suivant s'applique sur les données de migrations résidentielles (changements de domicile) produites par l'Insee, données contenues dans le fichier MobResid08.txt. Ce fichier est chargé avec la fonction read.csv2() qui lit un fichier texte selon les conventions françaises (cf. Section 2.2.3Codage des caractères et des séparateurs) : le point-virgule comme séparateur de colonnes et la virgule comme séparateur décimal.
2.
3.
4.
5.
residFlows <- read.csv2("data/MobResid08.txt",
stringsAsFactors = FALSE)
colnames(residFlows)
## [1] "CODGEO" "LIBGEO" "DCRAN" "L_DCRAN" "NBFLUX"
Les colonnes CODGEO et LIBGEO donnent le code et le nom de la commune d'origine de la migration résidentielle, les colonnes DCRAN et L_DCRAN donnent le code et le nom de la commune de destination et la colonne NBFLUX donne le nombre de migrants correspondant à chaque couple origine-destination. Il s'agit donc ici d'un format long que l'on souhaite transformer en format wide. Le tableau de données pourrait être transformé directement avec dcast() mais il est plus sûr d'utiliser d'abord melt() pour déclarer les identifiants et les mesures.
2.
3.
4.
5.
6.
7.
8.
meltedFlows <- melt(residFlows,
id.vars = c("CODGEO", "DCRAN"),
measure.vars = "NBFLUX")
odMatrix <- dcast(meltedFlows, CODGEO ~ DCRAN)
dim(odMatrix)
## [1] 143 144
On obtient ainsi une matrice origine-destination de 143 lignes par 143 colonnes qui stocke tous les flux résidentiels entre les 143 communes de Paris et de petite couronne. Une 144e colonne est créée qui contient les identifiants des communes : pour ne conserver que les flux, cette colonne pourrait être supprimée, à condition de conserver les identifiants en tant que noms de lignes avec la fonction row.names().
2-5. Packages polyvalents▲
Ce chapitre a présenté les fonctions les plus utiles pour manipuler des données, en particulier des tableaux de données : sélections, tris, recodages, superpositions et jointures. La plupart de ces fonctions font partie du tronc commun de R (r-base). Il existe cependant deux packages polyvalents qui permettent d'effectuer ces opérations et qui présentent de nombreux avantages : sqldf et dplyr.
Le package sqldf contient une seule fonction éponyme avec laquelle il est possible d'exécuter des requêtes SQL. Le langage normalisé SQL (Structured Query Language) est très expressif et il est utilisé pour la manipulation des bases de données (sqlite, MySQL, LibreOffice Base, MS Access) ainsi que dans certains logiciels de statistiques (SAS) ou de cartographie (MapInfo). Les utilisateurs qui ont déjà une pratique du SQL pourront ainsi directement s'exprimer dans ce langage.
Voici un exemple simple qui consiste à faire une sélection de lignes et de colonnes : sélection des communes dont la superficie est supérieure à 1 500 ha et conservation de trois colonnes du tableau, le code de la commune, son nom et sa surface).
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
library(sqldf)
selecArea <- sqldf("select CODGEO, LIBELLE, SURF
from popCom3608
where SURF > 1500")
selecArea
## CODGEO LIBELLE SURF
## 1 75112 Paris 12e Arrondissement 1632
## 2 75116 Paris 16e Arrondissement 1637
## 3 93005 AULNAY-SOUS-BOIS 1576
## 4 93073 TREMBLAY-EN-FRANCE 2463
La langage SQL exécuté dans R est identique à celui exécuté dans les autres logiciels, c'est tout l'avantage de la normalisation de ce langage de requête. Il n'est pas décrit plus précisément ici et l'utilisateur peut se référer aux très nombreux manuels et tutoriels sur le sujet.
Le package dplyr est une ré-écriture récente (janvier 2014) du package plyr. Au moment d'écrire ces lignes, il est difficile d'évaluer l'impact qu'il aura sur la communauté d'utilisateurs de R mais il s'agit probablement d'un jalon majeur dans l'évolution du logiciel. Pour résumer, il permet de faire toutes les opérations décrites dans ce chapitre, sa syntaxe est plus simple et plus lisible, il est plus rapide et il résout bon nombre de problèmes liés à la manipulation de très gros jeux de données.
Problèmes de mémoire : à la différence des bases de données et des logiciels d'analyse classiques, tous les objets utilisés dans R sont chargés dans la mémoire vive de l'ordinateur. La gestion de la mémoire est donc un problème important avec R et constitue un frein pour les utilisateurs voulant manipuler des données massives. La solution la plus simple dans ce cas est de stocker les données dans une base de données externe et d'y accéder depuis R, tâche que le package dplyr rend simple et efficace.
Les fonctions principales proposées par ce package sont filter(), arrange(), select(), mutate(), group_by() et summarise() auxquelles il faut ajouter les fonctions dédiées au jointures, left_join(), inner_join(), etc. La plupart de ces fonctions peuvent être exécutées sur un tableau classique (data.frame) mais il est conseillé de passer par le format spécifique de dplyr, ce qui se fait très simplement grâce à la fonction tbl_df().
Quelques manipulations similaires à celles présentées plus haut avec les fonctions classiques sont répétées ici pour illustrer l'intérêt de dplyr. Dans un premier temps, les communes du département de Seine-Saint Denis (93) sont sélectionnées, elles sont ensuite triées en fonction de leur population en 2008 et un tableau final est créé qui ne contient que le nom de la commune et la population.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
13.
14.
15.
16.
17.
18.
19.
20.
library(dplyr)
##
## Attaching package: 'dplyr'
##
## The following objects are masked from 'package:plyr':
##
## arrange, desc, failwith, id, mutate, summarise,
summarize
##
## The following objects are masked from 'package:stats':
##
## filter, lag
##
## The following objects are masked from 'package:base':
##
## intersect, setdiff, setequal, union
popComTbl <- tbl_df(popCom3608)
selecDep93 <- filter(popComTbl, DEP == "93")
sortedPop93 <- arrange(selecDep93, POP2008)
finalTab <- select(sortedPop93, LIBELLE, POP2008)
Problèmes de redondance : en 2014, plus de 5 000 packages sont disponibles et certaines des fonctions proposées ont des noms identiques. Par défaut, si on charge un package qui contient une fonction déjà chargée, c'est la fonction nouvellement chargée qui masque la fonction précédente. C'est le cas ici avec les fonctions arrange() ou mutate() chargées plus haut avec plyr et chargée à nouveau avec dplyr. Pour être sûr de savoir quelle fonction est appelée, le nom du package peut être spécifié devant le nom de la fonction avec la syntaxe suivante (exemple) : plyr::arrange().
Le package dplyr propose une nouvelle syntaxe pour enchaîner une suite d'opérations. Comparez les deux options suivantes qui exécutent le même enchaînement que dans l'exemple précédent. Dans le premier cas, l'enchaînement se fait de l'intérieur vers l'extérieur (syntaxe classique) ; dans le second cas, l'enchaînement se fait de gauche à droite grâce à l'opérateur %>%, il est plus lisible puisqu'il correspond à l'ordre du raisonnement.
2.
3.
4.
5.
6.
7.
8.
9.
10.
finalTab <- select(
arrange(
filter(popComTbl, DEP == "93"),
POP2008),
LIBELLE, POP2008)
finalTab <- popComTbl %>%
filter(DEP == "93") %>%
arrange(POP2008) %>%
select(LIBELLE, POP2008)
Les deux autres fonctions principales de dplyr sont illustrées dans l'exemple suivant. Il s'agit de calculer la densité de population dans les communes en 2008 puis de calculer la densité moyenne par département.
2.
3.
4.
5.
6.
7.
8.
9.
10.
11.
12.
popComTbl %>%
group_by(DEP) %>%
mutate(DENS = POP2008 / SURF) %>%
summarise(AVGDENS = mean(DENS))
## Source: local data frame [4 x 2]
##
## DEP AVGDENS
## 1 75 233.42
## 2 92 111.10
## 3 93 74.33
## 4 94 65.26
La page web dédiée au package dplyr(8) contient plusieurs vignettes très didactiques pour aller plus loin.
Au terme de ce chapitre, l'utilisateur a toutes les cartes en main pour importer et exporter des données, pour les examiner et pour les manipuler. Il dispose d'un ensemble de fonctions permettant d'effectuer les opérations les plus fréquentes en toute sécurité.





